In Zusammenarbeit mit Joan Lunney (ARS, Landwirtschaftsministerium der Vereinigten Staaten) und Jack Dekkers (Iowa State University) wurde das PRRS Host Genetics Consortium (PHGC) eingerichtet, um genomische und genetische Instrumente einzusetzen, mit denen sich die Auswirkungen von PRRS verringern lassen. Der Ansatz kombiniert modernste Verfahren der Virologie und Immunologie sowie der Genetik und Genomik. Die Hauptleistung besteht in der Anwendung der Ergebnisse auf kommerzielle Zuchtprogramme. Frühere Versuche, Gene zu identifizieren, die zur Resistenz gegenüber Krankheiten führen, konzentrierten sich auf den Vergleich der Reaktion verschiedener reiner genetischer Linien von Schweinen auf die Krankheit. Das PHGC stellt keine Zuchtvergleiche an, sondern richtet sich auf die Identifizierung von Resistenz-/ Suszeptibilitätsmarkern, die in den Populationen von Handelstieren vorhanden sind, die verschiedene Krankheitsphänotypen zeigen. Nach diesem PHGC-Modell wurden experimentell 200 Ferkel mit gut gekennzeichneten PRRS-Viren infiziert. Wichtige phänotypische Krankheitsmerkmale, die für jedes Schwein erfasst wurden, beinhalten die Mortalität, die Viruslast im Serum, die Gewichtszunahme sowie die Konzentrationen der neutralisierenden Antikörper und die Cytokin-Konzentrationen im Serum (s. Abb. 1). Durch die Genotypisierung aller Schweine mit dem porzinen 60K-SNP-Chip und die Durchführung genomweiter Assoziationsstudien konnten verschiedene Marker identifiziert werden, die mit dem Fortschreiten der klinischen Erkrankung und der Resistenz verknüpft sind. Das vorteilhafte Allel an einem wichtigen Marker auf dem Sus scrofa Chromosom 4 (SSC4) wird mit erhöhter Gewichtszunahme und verringerter Viruslast in Verbindung gebracht. Im Verlauf der 42 Tage dauernden experimentellen Infektionsphase, nahmen die Schweine mit dem begünstigenden Genotyp ca. 10% mehr an Gewicht zu und zeigten eine entsprechende Verringerung der Virämie. Die Wirkung der vorteilhaften Genotypen ist in Abb. 2 aufgeführt. Die bisherigen Ergebnisse deuten darauf hin, dass ein ähnlicher Vorteil des begünstigenden Allels unter Feldbedingungen erzielt werden kann.

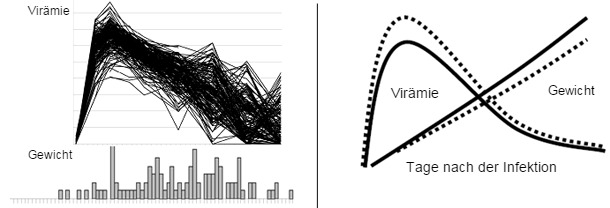
| Abb.1. PRRSV-Krankheitsphänotypen auf Populationsebene. Die obigen Abbildungen zeigen die PRRSV-Virämie (obere Abb.) und die Verteilung der Gewichtszunahme (untere Abb.) bei 200 Schweinen, die im Tierversuch mit PRRSV infiziert worden waren. Die Schweine wurden im Alter von 3 Wochen infiziert und die Daten 42 Tage lang nach der Infektion erhoben.. | Abb. 2. Der begünstigende Effekt der vorteilhaften Marker im SSC4 auf die Gewichtszunahme und die Virämie der Ferkel. Die obige Abbildung zeigt die erwarteten Fortschritte bei der Gewichtszunahme und Virämie der Schweine, die mit dem begünstigenden Allel (AB oder BB, durchgezogene Linie) ausgestattet sind, im Vergleich zu den Schweinen mit dem AA Genotyp (gestrichelte Linie). Der Anstieg der Gewichtszunahme liegt bei ca. 10%. Der Rückgang des Virämiehöhepunkts beträgt ca. 1 log. |
Zusätzliche phänotypische Auswirkungen werden mit verschiedenen Aspekten PRRSV-spezifischer Immunität in Verbindung gebracht. Wir stellten beispielsweise ein umgekehrtes Verhältnis zwischen der Viruslast und dem Virus-neutralisierenden Titer fest. Darüber hinaus ruft ein geringer Anteil infizierter Schweine eine einmalige Antikörperreaktion hervor, die als weitgehend neutralisierend beschrieben werden kann, verbunden mit der Kapazität, ein breites Spektrum von Isolaten, einschließlich Viren des Typs 1, zu neutralisieren. Obwohl für die neutralisierende Reaktion kein vererblicher Anteil festgestellt wurde, besitzt die Konzentration der anti-N proteinspezifischen Antikörper eine wichtige vererbbare Komponente und ist mit einem SNP auf SSC7 verbunden. Ein Marker auf SSC1 geht mit starken Atemwegserkrankungen bei Schweinen mit PRDC einher. Der nächste genomische Marker liegt in unmittelbarer Nähe eines Rezeptors für inflammatorische Zytokine.
Die Ergebnisse von Studien, die vom PHGC durchgeführt wurden, veranschaulichen wichtige Ergebnisse. Erstens unterliegen die klinische Erkrankung und die Folgen einer PRRS-Infektion in wesentlichem Maße erblichen Faktoren. Zweitens wird eine relativ geringe Anzahl an genomischen Markern mit einem bestimmten Phänotyp in Verbindung gebracht. Dies ermöglicht es, eine markergestützte Selektion zur Zucht von Schweinen mit verbesserten Antikörperreaktion auf Infektionen durchzuführen. Und schließlich befinden sich bisher alle genomischen Marker, die mit der klinischen Erkrankung und der Immunität verbunden sind, in Regionen des Schweinegenoms, die immunologisch relevante Gene, wie beispielsweise Interferon-stimulierte Gene, MHC-Gene und Zytokin-Antwort-Gene, umfassen. Schlussfolgernd sind also die Genomik und die Genetik neue Instrumente zur Verbesserung der Tiergesundheit, insbesondere für diejenigen ansteckenden Krankheiten, für die es keine wirksamen Impfstoffe oder Behandlungen gibt. Wenn Fortschritte in der Genetik mit besseren Impfstoffen und einer besseren Ernährung kombiniert werden, wird es uns möglich sein, viele der negativen Effekte endemischer PRRS umzukehren.