Eine umfangreiche Karriere in der PRRSV-Forschung
Dr. Hans Nauwynck, Professor an der Universität Gent (Belgien), kann auf eine beeindruckende und produktive Forscherkarriere zurückblicken. Er ist Autor von mehr als 450 fachlich geprüften Artikeln, 3 Buchkapiteln und 76 wissenschaftlichen Arbeiten, die er in den letzten 30 Jahren verfasste. Er gilt als einer der wichtigsten europäischen Forscher und ist mit Sicherheit der energischste! In diesem Artikel fassen wir unsere Diskussionen mit ihm darüber zusammen, wie sich PRRSV im Laufe der Zeit in den Betrieben weiterentwickelt hat.

Probleme bezüglich PRRSV Typ 1
Zu Beginn seiner Karriere (90er Jahre) führte er Experimente durch, um das Verhalten von PRRSV-1 und PRRSV-2 zu untersuchen. Obwohl beide Stämme zu reproduktiven Störungen bei Sauen führten, zeigten sie sich auf Ebene der Atemwege bei jungen Schweinen völlig unterschiedlich. Bei Typ 2 war Atemnot ein augenfälliges Symptom. Typ 1-Stämme führten vor allem zu subklinischen Infektionen. In diesen Jahren verstand er, dass wir es in der EU mit einem völlig anderen Virus zu tun hatten als in den USA und Kanada. Der starke genetische Unterschied stimmte mit den klinischen Befunden überein (Abb. 1). Auch die Felderfahrungen waren sehr unterschiedlich. Mit den PRRSV-1-Stämmen verbreitete sich das Virus in den Mastställen extrem langsam. Es dauerte eine ganze Mastperiode, bis das Virus alle Schweine infiziert hatte. Dies deutete auf ein sehr ineffizientes Übertragungsvermögen hin. Eine schnellere Ausbreitung wurde bei PRRSV-2 beobachtet. In den USA wiesen Wissenschaftler eine aerogene Ausbreitung nach. In den 00er Jahren wurden in Russland/Belarus genetisch abweichende PRRSV-1-Stämme (Subtypen 2 und 3) identifiziert, die wesentlich virulenter und pathogener waren. In jüngerer Zeit breitet sich PRRSV-1 Subtyp 1 schneller aus und verursacht sogar offenkundige Erkrankungen (italienische Stämme).
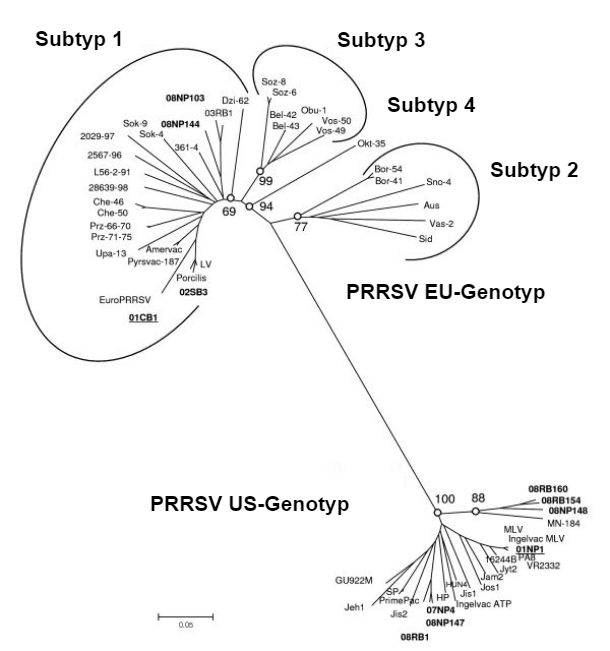
Abbildung 1: Phylogenetische Beziehung der PRRS-Viren, ORF5-Sequenzen, die den genetischen Unterschied zwischen PRRSV Typ 1 (EU-Genotyp) und PRRSV Typ 2 (US-Genotyp) verdeutlichen. Quelle: Amonsin, A., Kedkovid, R., Puranaveja, S. et al. (2009)
„In Westeuropa muss man sich keine Sorgen um PRRSV Typ 1 als Erreger von Atemwegserkrankungen machen, aber man muss die damit verbundenen Krankheiten oder Koinfektionen fürchten und beachten”, so Dr. Nauwynck. Er bezeichnete die PRRS-bedingten Atemwegserkrankungen in der EU als „PRRS PLUS“ (Diesen Begriff führte er für die Tierärzte ein, die in den Schweinebetrieben arbeiten) (Abb. 1). Die vielfältigen PRRSV-Koinfektionen mit verschiedenen anderen Erregern machen PRRSV zu einem der wichtigsten Krankheitserreger in der EU. Daher ist eine gründliche Diagnose unerlässlich. Alle Erreger sollten identifiziert werden. In diesem Zusammenhang benötigen wir eine neue Diagnostik. Eine vollständige Kenntnis der zirkulierenden Erreger ist wichtig um zu verstehen, was in dem Betrieb vor sich geht, und um in der Lage zu sein, PRRS PLUS zu kontrollieren. „Wenn die Tierärzte sich um alle potenziellen Koinfektionen kümmern, werden die klinischen respiratorischen Symptome von PRRS größtenteils verschwunden sein.“ Dr. Nauwynck glaubt, dass europäische positive Betriebe Schweine sehr effizient halten können, wenn sie die Kofaktoren kontrollieren, und das Virus nicht ausrotten müssen. Das kann sich natürlich ändern, wenn die Virulenz/Pathogenität des Virus in Zukunft zunimmt.
Die Evolution des Virus kann durch eine bessere Kenntnis der Interaktionen zwischen Virus und Wirt erklärt werden – die Rolle der Siglec-Rezeptoren
Das Team von Dr. Nauwynck konzentriert sich auf die Untersuchung der Virus-Wirt-Interaktion auf molekularer Ebene und konnte dadurch verstehen, wie sich das Virus im Laufe der Zeit verändert hat, insbesondere wie sich sein Übertragungsvermögen, wie oben beschrieben, und seine Pathogenität erhöhte.
Sein Team zeigte, dass der Viruseintritt in Makrophagen mehrere Schritte umfasst. Er beginnt mit der Interaktion des Virus mit Siglec-Rezeptoren, gefolgt von der Interaktion mit dem CD163-Rezeptor bei der Internalisierung, was zum Abbau führt. Verschiedene Siglecs können von PRRSV genutzt werden und haben sich im Laufe der Zeit entwickelt.
Man kann davon ausgehen, dass Siglec-10 ein „archaischer Rezeptor“ ist. Siglec-10 ist auf einer kleinen Untergruppe von Makrophagen in Tonsillen und anderen lymphatischen Geweben vorhanden und ist sehr effizient beim Eintrittsprozess des Virus, was seine lange Koevolution mit PRRSV beweist. Dieses Siglec wurde höchstwahrscheinlich ganz am Anfang der Entstehung des Virus benutzt. Zu dieser Zeit replizierte PRRSV nur in dieser kleinen Population von Makrophagen und verursachte höchstwahrscheinlich noch keine klinischen Symptome. Da Siglec-10 auch auf B-Lymphozyten vorhanden ist, konnte PRRSV zwar binden und in diese Zellen eindringen, aber diese nicht infizieren. Dies könnte die Grundlage für die Persistenz des Virus in seinem Wirt (Reservoir) sein. Ende der 80er Jahre wechselte PRRSV höchstwahrscheinlich zusätzlich auf den Siglec-1-Rezeptor (= Sialoadhesin, CD169) (Abb. 2). Siglec-1-positive Zellen sind in der Lunge, der Plazenta und den lymphatischen Geweben zu finden. Die Vermehrung in der Lunge und der Plazenta führte zu respiratorischen und reproduktiven Problemen. Daher der Name PRRSV. In den 00er Jahren begann das Virus in Westeuropa, sich in den nasalen Makrophagen zu replizieren, was zu einer Zunahme der Übertragung zwischen Schweinen führte. Der Rezeptor, der für diese Anpassung verantwortlich ist, ist nicht bekannt, aber er unterscheidet sich von Siglec-1 und Siglec-10. Der hochvirulente Lena-Stamm (PRRSV-1 Subtyp 3) führt zu Gefäßerkrankungen, die mit der Replikation von PRRSV in venösen Makrophagen in Verbindung stehen. Der Rezeptor in dieser Untergruppe von Makrophagen unterscheidet sich ebenfalls von Siglec-1 und Siglec-10. Die Rezeptoren in den nasalen und venösen Makrophagen werden derzeit untersucht.
Dr. Nauwynck glaubt, dass sich das Virus zu Formen weiterentwickelt, die immer mehr Subpopulationen von Makrophagen infizieren.
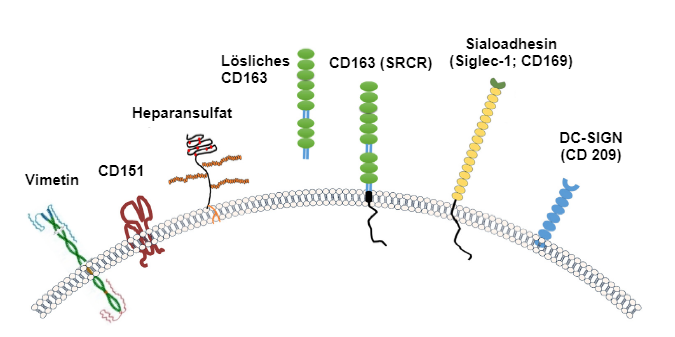
Abbildung 2: Darstellung der allgemein für PRRSV in Betracht kommenden Rezeptoren. Quelle: Zhang, Q., Yoo, D. (2015)







