Die Diversität von Influenzaviren lässt sich hauptsächlich durch zwei Faktoren erklären, die eng miteinander verbunden sind: auf der einen Seite die evolutionären Mechanismen dieser Viren – Gendrift und Reassortment (die bereits in einem anderen Artikel dieser Rubrik besprochen wurden) – und auf der anderen Seite das epidemiologische Verhalten des Virus bei jeder Spezies, was erhebliche Auswirkungen auf die evolutionären Mechanismen hat.
Wenn wir die Evolution dieser bei Schweinen vorkommenden Viren bezüglich der Gendrift mit den derzeit beim Menschen vorkommenden Viren vergleichen, sollten zwei Phänomene hervorgehoben werden: 1) Bezüglich eines bestimmten Stamms variiert das Virus antigenisch schneller beim Menschen als beim Schwein, obwohl die genetischen Mutationsraten bei beiden Arten gleich sind, und 2) weltweit ist die Diversität der Influenzaviren auf genetischer und antigenischer Ebene beim Schwein höher als beim Menschen. Obwohl diese beiden Tatsachen auf den ersten Blick widersprüchlich erscheinen mögen, sind sie vollkommen kompatibel, wie wir im Folgenden sehen werden.

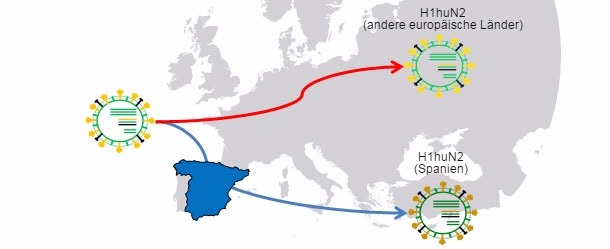
Abbildung 1: Gemeinsamer Vorgänger von H1huN2 (1994); Geografische Isolation → Drift führt zu länderbezogenen Varianten
Wie wir in früheren Veröffentlichungen gesehen haben, kann die Grippe in Schweinebetrieben als eine enzootische Krankheit zirkulieren, d. h. das Influenzavirus kann sich in Betrieben ansiedeln. Diese Situation findet sich sehr häufig und ist mit dem Auftreten von wiederkehrenden Zirkulationen verbunden (Loeffen et al., 2009, Simon-Grifé et al., 2012, Rose et al., 2013). In einer Veröffentlichung von 2001 (de Jong et al.) wurden mehr als 20 avian-like H1N1-Isolate (H1avN1) in genetischer und antigenischer Hinsicht miteinander verglichen. Diese Viren wurden in den Niederlanden bei 6 Ausbrüchen endemischer Schweineinfluenza isoliert, die in Betrieben aufgetreten waren. Bezüglich des Hämagglutinin-Gens (H) häuften sich diese Viren in fünf Gruppen, deren Geno- und Phänotyp sich voneinander unterschieden. Dabei ist darauf hinzuweisen, dass jede Gruppe einem Betrieb zugeordnet werden konnte (abgesehen von einer Gruppe, die Isolate aus zwei Betrieben aufwies). Diese Studie belegt, dass alle H1avN1-Isolate einen gemeinsamen Vorgänger hatten, das endemische Zirkulieren in jedem Betrieb aber die Diversifizierung in fünf Cluster durch Drift forcierte. In einer anderen Studie (Martin-Valls et al., 2013), die zwischen 2009 und 2012 am CReSA durchgeführt wurde, wurden 4 H1N2-Viren isoliert. Bei der Analyse wurde festgestellt, dass H1 aller Isolate zum „menschlichen“ H1N2-Typ gehörte (H1huN2, häufigster europäischer Stamm der Schweineinfluenza), sich aber eine eigene Gruppe innerhalb des H1huN2-Clusters ausdifferenzierte. Deshalb kann man davon ausgehen, dass hier eine Evolution und Differenzierung stattfand, die durch die geografische Isolation in Spanien forciert wurde. Später stellten wir fest, dass diese Viren eine eingeschränkte Reaktion mit Seren zeigten, die mit hohen Titern auf handelsübliches H1huN2 reagiert hatten, was auf Antigendrift hindeutet (unveröffentlichte Daten). Kurz gesagt tritt Gen- und Antigendrift in Betrieben in endemischen Situationen und bei geografischer Isolation auf. Diese Phänomene spielen eine wichtige Rolle beim Auftreten neuer antigenischer Varianten. Beim Menschen ist die Situation jedoch ganz anders. In diesem Fall könnte man sagen, dass sich die Viren aufgrund des gesamten Immundrucks antigenisch ändern müssen um zu überleben. Derzeit verhindern die zahlreichen und vielfältigen Verbindungen zwischen Ländern der ganzen Welt Situationen viraler Isolation. Deshalb gibt es beim Menschen eine insgesamt positive Selektion jener Varianten der Influenzaviren, die fähig sind, sich der Immunantwort zu entziehen. Dieses Phänom führt zur Notwendigkeit, die Grippeimpfstoffe für den Menschen ständig anzupassen.
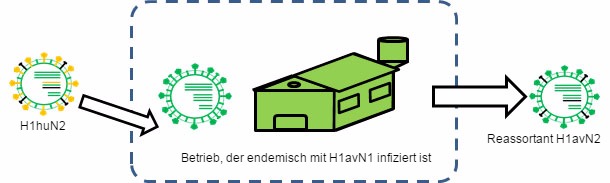
Abbildung 2: H1huN2 wird in einen Betrieb eingeschleppt, der endemisch mit H1avN1 infiziert ist. → Reassortment tritt auf.
Reassortment kann auch eine Quelle antigenischer Veränderungen sein. Beim Menschen beispielsweise wurde das Reassortment zu einer der Hauptursachen der letzten drei Pandemien. Die pandemischen Viren waren Reassortanten, die mindestens ein neues Hämagglutinin beinhalteten, das sich antigenisch unterschied. Da die menschliche Bevölkerung gegenüber diesen neuen reassortierten Viren immunologisch naiv war, traten Übertragungen auf pandemischer Ebene auf. Wie wir bereits in einem anderen Artikel dieser Rubrik gesehen haben, treten Reassortments der Grippeviren bei Schweinen häufig auf und ein großer Teil der Schweineinfluenza-Stämme ist durch diese evolutionären Mechanismen entstanden. Beispiele hierfür sind H1avN2, ein H1N2-Virus, das mit dem H1avN1-Virus kreuzreagierte (Rose et al., 2013) oder H1huN1, das sich antigenisch mit H1huN2 kreuzte (Martín-Valls et al., 2013, Rose et al., 2013). Damit ein Reassortment stattfinden kann, sind zwei Stämme notwendig, die dasselbe Tier zur selben Zeit infizieren. Offensichtlich sind endemische Situationen ideale Voraussetzungen dafür. Bei der Beurteilung des Auftretens der endemischen Grippe gab es tatsächlich Hinweise auf das Reassortment zwischen verschiedenen Stämmen und sogar Subtypen (Martin-Valls et al, 2013; Rose et al., 2013).
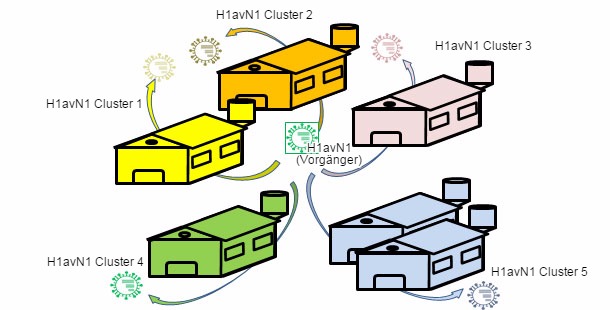
Abbildung 3: Endemische Infektionen erleichtern die isolierte Evolution durch Drift in jedem Betrieb. → Betriebsbezogene genetische und antigenische Heterogenität
Abschließend lässt sich sagen, dass Schweineinfluenzaviren eine hohe genetische und antigenische Diversität aufweisen. Diese Diversität ist das Ergebnis der spezifischen evolutionären Mechanismen dieser Viren in Kombination mit epidemiologischen Faktoren wie z. B. der endemischen Zirkulation innerhalb eines Betriebs und/oder der geografischen Isolation. Das Zusammenwirken all dieser Faktoren kann eine fehlende Immunerkennung zur Folge haben, was zu eingeschränkter Kreuzreaktivität bei Viren führt, die einen gemeinsamen Vorgänger haben oder zum gleichen Subtyp gehören.



