ELISA und PCR sind die zwei am häufigsten verwendeten Diagnoseverfahren zum Nachweis von PRRS. In diesem Artikel werden wir uns auf PCR konzentrieren, dem Verfahren, mit dem das Virus in praktisch jedem Material nachweisbar ist. Es beruht auf einer zyklischen Polymerisation einer DNA oder wie im Fall von PRRSV eines cDNA-Fragments, das von kurzen Sequenzen flankiert wird, an die sich in jedem PCR-Zyklus spezielle DNA-Primer binden. Die Sensitivität und Spezifität eines diagnostischen, PCR-basierten Tests hängt von der Eignung dieser Primer bezüglich ihres Ziels und den Thermozyklen ab, die für die zyklische Amplifikation benötigt werden. Idealerweise sollten sie nur komplementär zum Genom einer bestimmten Virusart und nicht in der Lage sein, sich an eine andere Mikrobe zu binden oder DNA-Sequenzen zu enthalten, die noch Genome aller Stämme oder Varianten dieser Virusart erkennen können. Sie zielen also auf die am häufigsten erhaltenen Fragmente des Genoms ab.
PRRSV ist einer der schwierigsten Krankheitserreger, wenn es um die Entwicklung hochsensibler und spezifischer PCR-Assays geht. Die derzeit bekannten PRRS-Viren gehören zu den zwei Arten PRRSV-1 und PRRSV-2. Beide PRRSV-Spezies sind weltweit verbreitet, aber PRRSV-1 dominiert in Europa, während PRRSV-2 in Amerika und Asien vorherrscht. Man kann davon ausgehen, dass die genetische Vielfalt einer dominierenden PRRSV-Spezies höher ist als diejenige, die eine geringere Prävalenz hat. In der Tat können PCR-Assays, die zum Beispiel in Europa oder in Amerika zum Einsatz kommen, für den Nachweis von PRRSV-1 bzw. PRRSV-2 eine bessere Validität haben und besser geeignet sein. In der Folge bleiben einige Stämme der „unbedeutenderen“ Arten in einem bestimmten Gebiet oder einem Kontinent möglicherweise unerkannt, womit ihre Rolle bei der PRRS-Pathogenese unterschätzt werden könnte. Das ist besonders wichtig in Betrieben, in denen PRRSV-1 und PRRSV-2 nebeneinander zirkulieren. Die große genetische Vielfalt der PRRS-Viren erfordert eine ständige Aktualisierung der Sequenzen von PCR-Primern und -Sonden, um die kontinuierliche Entwicklung des Virus und die Entdeckung völlig neuer genetischer Varianten zu verfolgen. Dies gilt insbesondere für PRRSV-1, dessen extreme genetische Vielfalt und geringe Verfügbarkeit von Referenzviren aus Russland, Weißrussland und der Ukraine eine Gefahr für die beeinträchtigte Sensitivität kommerzieller PCR-Kits gegenüber den osteuropäischen genetischen Subtypen von PRRSV-1 darstellt.

Derzeit wird die Variante der Realtime-PCR meist in der Routinediagnostik eingesetzt. Es gibt mehrere Realtime-PCR-Verfahren, die sich in ihren Grundsätzen unterscheiden, aber das am weitesten verbreitete ist ein Verfahren mit sogenannten TaqMan-Sonden. Eine TaqMan-Sonde ist ein kurzes DNA-Fragment (Oligonukleotid), das mit Fluorophoren markiert wurde und die Ziel-DNA zwischen den flankierenden Primern bindet. Wenn eine Bindung erfolgt, wird die Sonde während der DNA-Polymerisation lysiert und die Fluoreszenz wird vom PCR-Block registriert. Die Realtime-PCR eignet sich besonders gut für das Multiplexing der diagnostischen Ziele. Dies bedeutet, dass in einer einzigen PCR-Reaktion (oder einem einzigen Test) 2-3 verschiedene Krankheitserreger durch die Emission von Fluoreszenz von 2-3 verschiedenen Farbstoffen nachgewiesen werden können. So lassen sich gleichzeitig beide PRRSV-Arten leicht erkennen und in einer Stichprobe von einem einzigen Tier, einer Bucht oder einem Betrieb unterscheiden.
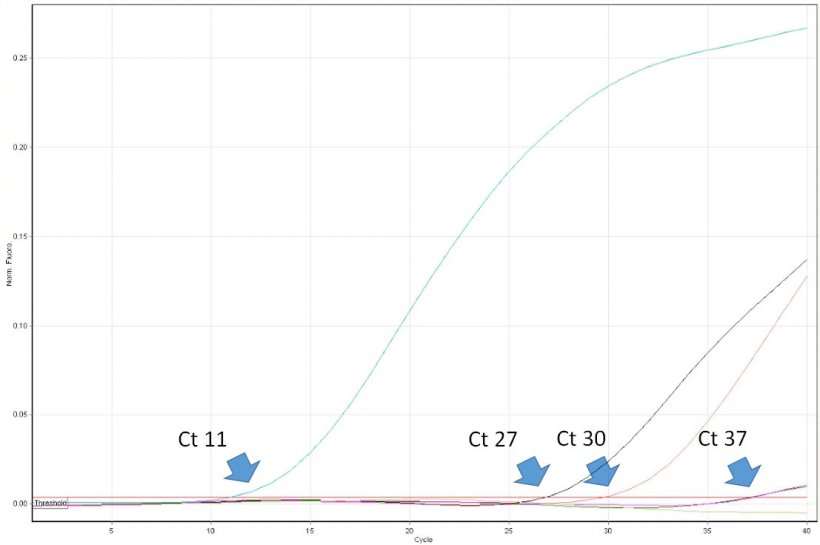
Abbildung 1: Der Schwellenwertzyklus (Ct-Wert) ist die erste Zyklusnummer der quantitativen Echtzeit-PCR, bei der Fluoreszenz festgestellt wird, was auf das Vorhandensein des entsprechenden Erregers in der Probe hinweist. Je niedriger der Ct-Wert, desto höher ist die Menge des Krankheitserregers in der getesteten Probe. Sehr hohe Ct-Werte sind mit Vorsicht zu interpretieren, da sie trotz fehlender Ziel-DNA in der Probe aus dem spontanen Zerfall einer TaqMan-Sonde in den sehr späten Zyklen entstehen können.
Die Interpretation der PCR-Ergebnisse ist trügerisch leicht. Tierärzte müssen sich einiger Einschränkungen des Verfahrens bewusst und in der Lage sein, die Ergebnisse kritisch zu interpretieren. Das positive Ergebnis von PCR weist beispielsweise das Vorhandensein einer Zielnukleinsäure (z. B. von PRRSV) in der Probe nach. Es belegt aber nicht, dass die Stichprobe infektiöse Viren enthielt. Die Realtime-PCR ermöglicht die Quantifizierung des Virus in der Probe. Ihre Ergebnisse werden als „Schwellenwertzyklus“ (Ct-Wert) angegeben. Es ist die erste Zyklusnummer, bei welcher der PCR-Block die Fluoreszenzemission aus dem Reaktionsröhrchen erkannte. Je niedriger die Zykluszahl ist, umso höher ist die Kopienzahl der Ziel-DNA (zu verstehen als Zahl von Viruskopien). Beispielsweise kann eine Probe mit einem Ct-Wert von 20 als sehr positiv angesehen werden und eine Probe mit einem Ct-Wert von 35 als gering positiv. In einigen Fällen wird die Fluoreszenz jedoch nur in sehr späten Zyklen der Realtime-PCR, z. B. bei > 38, festgestellt. Wie lassen sich solch hohe Werte interpretieren? Leider deutet ein solches Ergebnis nicht immer auf das Vorhandensein einer sehr geringen Anzahl von Viruskopien in der Probe hin. Die sogenannten „Late-Riser“ können das Ergebnis eines spontanen Verfalls einer Sonde nach vielen Thermozyklen der PCR sein, die enzymatische Nukleolyse durch DNA-Polymerase imitieren. Dies kann somit auch bei nicht vorhandener Nukleinsäure des Virus in der Probe auftreten. Die Bedeutung der korrekten Interpretation von „Late-Rrisern“ ist bei der Überwachung der PRRSV-freien Populationen am größten. Eine Follow-up-Diagnose, einschließlich ELISA, muss durchgeführt werden. Leider liefern einige Diagnoselabore in Europa den Kunden keine präzisen Ct-Werte der Realtime-PCR. Stattdessen werden die Ergebnisse nur als negativ oder positiv angegeben. Die Nützlichkeit solcher Berichte für Tierärzte ist begrenzt und kann in einigen Fällen zu falschen Entscheidungen bezüglich der Protokolle zur PRRSV-Kontrolle führen.
Praktische Fragen
- Keine der aktuellen PCR-Methoden ist in der Lage, alle PRRS-Viren zu erkennen.
- Das negative PCR-Ergebnis von ein paar Stichproben eines Betriebs beweist nicht, dass er frei von PRRSV-Infektionen ist.
- Ein positives PCR-Ergebnis weist das Vorhandensein einer PRRSV-Nukleinsäure in einer Probe nach, aber nicht unbedingt ihre Infektiosität.








