Klinische Symptome einer Infektion mit dem PRRS-Virus (PRRSV) können vielfältig sein, weshalb die klinische Diagnose durch Laboruntersuchungen zu bestätigen ist. Meistens werden ELISA und RT-PCR zum Nachweis von PRRSV-spezifischen Antikörpern bzw. von viralen Nukleinsäuren eingesetzt.
Die Wahl des geeigneten Diagnoseverfahrens hängt vom Diagnoseziel ab. Bei der Überwachung negativer Betriebe sind hochspezifische Methoden vorzuziehen, während die Untersuchung von Sperma einzelner Eber maximale Sensitivität und Methoden erforderlich macht, durch die eine Infektion zum frühestmöglichen Zeitpunkt festgestellt werden kann. Bei der Planung und Ausführung von Programmen zur PRRS-Bekämpfung ist der beste Ansatz die Kombination von ELISA und PCR-Methoden mit anschließender DNA-Sequenzanalyse. Das Probenahmeprotokoll in einem bestimmten Betrieb muss je nach Diagnoseziel und Organisation des Betriebs individuell geplant werden. Die ganz einfache Frage, ob ein Betrieb mit PRRSV infiziert ist, kann durch einen ELISA-Test von 10-20 Mastschweinen beantwortet werden. Eine detaillierte Analyse der PRRSV-Zirkulation in einem Betrieb mit Tieren aller Altersklassen vom Ferkel bis zum Mastschwein oder einem Multisite-Betrieb erfordert jedoch die Untersuchung einer großen Anzahl an Proben, die verschiedene Altersgruppen und alle Standorte des Produktionssystems repräsentieren, da sie verschiedene, voneinander unabhängige virale Ökosysteme abbilden können.

Die Interpretation einer Serokonversion oder der Nachweis des Virus in Betrieben, in denen modifizierte Lebendimpfstoffe zum Einsatz kommen, stellt eine Herausforderung dar, da (1) es keine Markerimpfstoffe auf dem Markt gibt, (2) Impfviren in geimpften Betrieben fortbestehen können und (3) sie zumindest einige Zeit neben Wildtyp-Stämmen existieren können. In diesen Betrieben ist eine DNA-Sequenzanalyse von Amplikonen erforderlich, die dort in jeweils verschiedenen Altersklassen erhalten werden, um die PRRSV-Zirkulation vollständig zu verstehen.
Die serologische Diagnose auf Grundlage von ELISA ist leicht durchzuführen und besitzt bei der Durchführung auf Betriebsebene grundsätzlich eine gute Spezifität und Sensitivität. Das Serum von einzelnen, insbesondere erwachsenen Schweinen ergibt jedoch manchmal falsch positive Reaktionen. Dies stellt ein Problem bei der Überwachung negativer Bestände dar und erfordert eine erneute Untersuchung der Proben mit anderen Methoden (z. B. IPMA, IFA oder einem anderen ELISA-Test mit höherer Spezifität) oder eine erneute Probenahme im Schweinebestand nach 2–3 Wochen. Dieser Aspekt der ELISA-Ergebnisse wird bei der Bewertung der diagnostischen Kapazitäten des Verfahrens selten berücksichtigt. Die geringe Sensitivität eines bestimmten ELISA-Kits kann dadurch kompensiert werden, dass man die Zahl getesteter Proben erhöht, während eine geringe Spezifität immer den Einsatz zusätzlicher Methoden erforderlich macht.
Generell eignet sich jedes ELISA-Kit für den Nachweis von Antikörpern gegen jeden der beiden PRRSV-Genotypen, aber Kits, die nur Antigene eines PRRSV-Genotyps verwenden, sind häufig weniger sensitiv gegenüber dem zweiten Genotyp. Es gibt auch Kits zur Unterscheidung der Serokonversion gegenüber beiden Genotypen, aber aufgrund der serologischen Kreuzreaktion zwischen den Genotypen sind die Ergebnisse mit Vorsicht zu interpretieren. Die unterscheidende PCR ist immer zu empfehlen, um die Diagnose der Doppelinfektion eines Betriebs mit zwei Genotypen zu bestätigen.
Die PCR-Methoden weisen virale Nukleinsäuren auf Grundlage der Komplementbindung kurzer synthetischer Oligonukleotide (Primer) an ein spezifisches Fragment des viralen Genoms nach, das später in einer enzymatischen Reaktion amplifiziert wird. In den meistbenutzten Realtime-PCR-Verfahren wird das Produkt einer Reaktion durch Überwachung eines Signals nachgewiesen, das von der DNA-Sonde nach der Bindung einer spezifischen Sequenz abgegeben wird. Aufgrund der Art dieser Reaktion kann ein solches Signal auch ohne eine virale Sequenz abgegeben werden, insbesondere bei den letzten PCR-Zyklen, was einem schwachen positiven Ergebnis ähneln kann.


Ellingson 2013, thesis, Iowa SU
Die größten Bedenken hinsichtlich der RT-PCR-Verfahren beziehen sich jedoch auf die hohe genetische Vielfalt des Virus, was zur Akkumulation von Nukleotid-Mutationen in den Genomfragmenten, in denen Primer und Sonden binden, und folglich zu falsch negativen PCR-Ergebnissen führen kann. Ergebnisse einer vergleichenden Analyse handelsüblicher RT-PCR-Kits, die von einem OIE-Referenzlaboratorium durchgeführt wurden (Podgorska et al., 2015), wiesen darauf hin, dass einige handelsübliche RT-PCR-Kits bei vielen PRRSV Typ 1 Stämmen, die hauptsächlich aus Osteuropa stammten, versagten. Aufgrund der beschränkten Verfügbarkeit von Sequenzen aus Osteuropa wurden die meisten Primer und Sonden bei derzeit benutzten Verfahren auf Grundlage der klassischen Stämme von Typ 2 und Typ 1 Subtyp 1 entwickelt, während sich osteuropäische Stämme der Subtypen 2-4 genetisch sehr stark unterscheiden (Stadejek et al. 2013). Überdies erwiesen sich einige von ihnen im Vergleich zu Stämmen, die in West- und Zentraleuropa zirkulieren, als virulenter, so dass man mehr Aufmerksamkeit auf sie richten sollte um im Falle ihre Ausbreitung eine frühe Erkennung sicherzustellen.
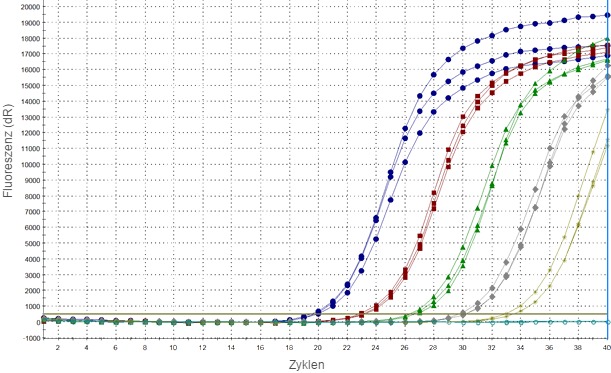
Derzeit kann keine einzige RT-PCR als universelle Methode empfohlen werden, die den Nachweis aller PRRSV-Stämme mit optimaler Sensitivität ermöglichen würde. Diese Beobachtungen unterstreichen die Notwendigkeit einer gründlichen Bewertung der derzeit benutzten Verfahren und ihrer ständigen erneuten Prüfung bezogen auf die aktuell zirkulierenden Stämme. Hierfür muss eine Sammlung repräsentativer PRRSV-Stämme erstellt werden, die die bekannte genetische Vielfalt des Virus abdeckt, was derzeit eines der wichtigsten Ziele der Referenzlaboratorien für PRRS darstellt.








