Infektionen mit dem Influenza-A-Virus (IAV) führen auf der ganzen Welt zu hoher Morbidität in Schweinebeständen (Brown, 2000). Die Infektion steht vor allem in Zusammenhang mit akuten Atemwegsbeschwerden, die häufig von bakteriellen Koinfektionen begleitet sind und im Endstadium zum Tod und erhöhter Letalität unter Saugferkeln, reduzierter Gewichtszunahme bei Mastschweinen oder zu Aborten bei tragenden Sauen führen können. Folglich können die wirtschaftlichen Schäden für die Besitzer ziemlich hoch sein (Rajao et al., 2014; Vincent et al., 2014; Vincent et al., 2014).
Porzine Influenza-A-Viren gehören zur Familie der Orthomyxoviridae. Es sind einzelsträngige RNA-Viren, die aus acht Gensegmenten bestehen. Die Subtypen werden entsprechend der genetischen und antigenischen Merkmale des Oberflächenglykoproteins Hämagglutinin (HA) und der Neuraminidase (NA) klassifiziert (Cheung et al., 2007).

Glykan-Gruppen mit endständigen Sialinsäuren agieren als Rezeptoren für die Anheftung des IAV an der Zelloberfläche im Respirationstrakt. Aufgrund der Expression der Rezeptoren mit α-2-6 oder α-2-3-gebundenen endständigen Sialinsäuren in den oberen Atemwegen des Schweins sind die Tiere anfällig für IAV aviären ebenso wie menschlichen Ursprungs (Byrd-Leotis et al., 2014; Trebbien et al., 2011; Walther et al., 2013). Deshalb eröffnen Koinfektionen porziner Zellen mit aviärem und/oder humanem IAV Möglichkeiten für Reassortments (Austausch von Genomsegmenten) zwischen diesen Viren (Hass et al., 2011; Ma et al., 2009; Van Poucke et al., 2010; Zell et al., 2013). Solche Reassortanten können unerwartete und schwer vorhersehbare phänotypische Veränderungen im Vergleich zu ihren parentalen Viren aufweisen, die beispielsweise den Wirts- und Gewebetropismus, die Virulenz und die antigenischen Eigenschaften betreffen.
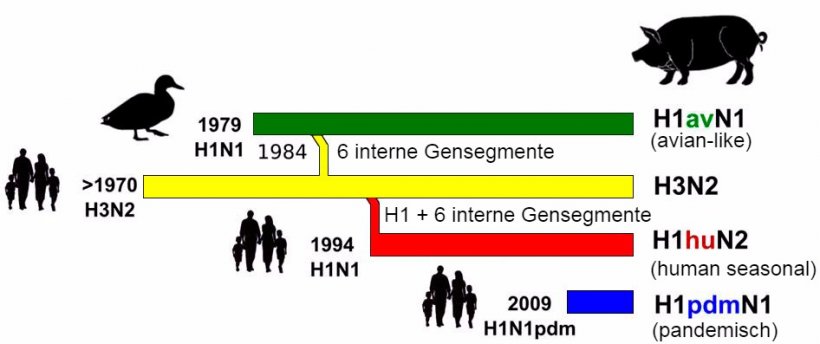
Abbildung 1: Geschichte und Ursprung porziner Influenza-A-Viren (IAV), die derzeit in Europa zirkulieren. Es ist festzustellen, dass keines dieser porzinen Influenza-A-Viren aus Schweinebeständen stammte.
Abbildung 1 gibt einen Überblick über die Herkunft und die Reassortierungsgeschichte der porzinen IAV, die derzeit in Schweinebeständen in Europa zirkulieren. Bis 2009 zirkulierten beim Schwein in Europa drei Subtypen von IAV: H1N1, H1N2 und H3N2. Der älteste dieser Virenstämme, H1N1, wurde Ende der 1970er Jahre aus einer aviären Quelle in die Schweinepopulation eingeschleppt und ersetzte den „klassischen” humanen H1N1-Virusstamm von 1918, der in den Jahren davor zirkulierte. Da es sich um ein Influenzavirus aviären Ursprungs handelt, wird der nach den 1970er Jahren auftretende H1N1-Virusstamm als „avian-like” (av) H1N1 bezeichnet (Brown, 2013; Simon et al., 2014). 1984 reassortierte ein humanes saisonales H3N2-Virus mit dem H1avN1av-Subtyp und brachte eine Reassortante hervor, die alle sechs internen Segmente aviären Ursprungs behielt und das HA (H3) und die NA (N2) des humanen Virus trug (Kong et al., 2015). Diese Reassortante wurde seither in Schweinebeständen in manchen, aber offensichtlich nicht in allen europäischen Ländern endemisch. Ein weiteres Reassortment betraf 1994 das porzine H3N2-Virus, das sein HA-Gensegment mit demjenigen eines anderen humanen saisonalen H1N1-IAV austauschte und den porzinen H1N2-Virusstamm hervorbrachte. Dieser H1-Subtyp wird als „human-like” (hu) H1N2 bezeichnet (Brown, 1998). Diese Virenstämme zirkulierten mit einigen geographischen Einschränkungen bezüglich ihrer Prävalenz mit relativer Stabilität in europäischen Ländern (Brown, 2013; Kuntz-Simon et al., 2009). Die Viren des H1N1av-Stamms schienen in Europa für die meisten Infektionen beim Schwein verantwortlich zu sein (Brown, 2000).
2009 kam mit dem humanen pandemischen H1N1 2009 Influenza-A-Virus (H1N1pdm) ein vierter Subtyp ins Spiel. Dieses Virus ist aus mehreren Reassortments hervorgegangen und besteht aus Segmenten des amerikanischen aviären (PB2, PA), des humanen (PB1), des klassischen porzinen (HA, NP, NS) und des europäischen porzinen Stamms (NA, NP) (Neumann et al., 2009). In Bezug auf das letzte humane pandemische IAV, das mindestens teilweise porzinen Ursprungs war, nannte man die Infektion beim Menschen „Schweineinfluenza”. Die Schweine schienen sehr anfällig für diese Art des IAV zu sein und die reverse zoonotische Übertragung von infizierten Menschen auf das Schwein wurde an vielen Orten weltweit dokumentiert. Seither verbreitet sich H1N1pdm in den Schweinebeständen und -zyklen unabhängig von Infektionen beim Menschen (Brookes et al., 2010).
Infolge dieser sogenannten „Schweineinfluenza” wurde 2013-2015 ein europäisches Überwachungsprogramm für die Influenza beim Schwein, das Projekt ESNIP3, durchgeführt (Watson et al., 2015). Die Ergebnisse zeigten, dass H1N1pdm in verschiedenen europäischen Ländern und mit besonders hoher Prävalenz in Großbritannien und Irland vorkommt. Das Vorkommen von H1N1pdm menschlichen Ursprungs in Schweinebeständen in Europa störte offensichtlich das ausgewogene Gleichgewicht der drei ursprünglich europäischen porzinen Influenzavirusstämme und führte zu vermehrten Reassortments. Unter den Reassortanten, die H- und/oder N-Segmente betrafen, tauchte ein H1pdmN2-Stamm in Deutschland auf, der inzwischen seit mehreren Jahren mit niedriger Prävalenz zirkuliert (Starick et al., 2012; Watson et al., 2015). Das HA-Protein dieser Reassortanten hat bedeutende Änderungen erfahren und kann antigenisch vom echten H1pdm menschlichen Ursprungs unterschieden werden (Harder et al., 2013). Andere Reassortanten (H1huN1av, H1dpmN1av, H3N1pdm) wurden beschrieben, konnten die Zirkulation aber offensichtlich nicht aufrechterhalten (Watson et al., 2015). Darüber hinaus wurde von Reassortanten berichtet, die alle oder einen Teil der sechs internen Genomsegmente mit H1N1pdm-Viren ausgetauscht haben (Kong et al., 2015; Watson et al., 2015). In den USA wurden porzine H3N2- oder H1N1-Viren, die M-Gensegmente von H1N1pdm trugen, in Verbindung mit einer verstärkten Übertragung vom Schwein auf den Menschen bei Landwirtschaftsmessen gebracht (Schicker et al. 2016). Von solchen Virusvarianten wurde auch sporadisch aus Europa berichtet, sie standen aber in keinem Zusammenhang mit Infektionen beim Menschen. Die Prävalenz von H1N1pdm unter Schweinebeständen in Europa variiert stark (Abb. 2). Im Gegensatz zur Situation auf den Britischen Inseln weist der porzine Subtyp H1N1av immer noch die höchste Prävalenz auf dem europäischen Kontinent auf, während für die Subtypen H3N2 und H1huN2 eine geringere Prävalenz festgestellt wurde (Abb. 2). Der Subtyp H3N2 wurde in Dänemark, Polen oder Großbritannien nicht festgestellt und es wurde nur von sehr wenigen, sporadisch auftretenden Ausbrüchen aus Frankreich berichtet (Watson et al. 2015).

Abbildung 2: Geographische Beschränkung der HA-Subtypen porziner Influenzaviren, die derzeit in Europa zirkulieren. Die Daten beruhen auf Literaturhinweisen und unseren eigenen Studien passiver Überwachung, mit denen 2015 begonnen wurde.
Studien zur passiven Überwachung in verschiedenen mittel- und westeuropäischen Ländern begannen nach Beendigung des ESNIP3-Projekts 2015 und wurden von unserem Labor durchgeführt. Vorläufige Daten, die im ersten Jahr der Überwachung gesammelt wurden, zeigten eine zunehmende Prävalenz von H1N1pdm und seinen Reassortanten. Außerdem waren die erhöhte Häufigkeit von Koinfektionen und ein vermehrtes Auftreten von Reassortments zwischen den drei zusammen zirkulierenden porzinen europäischen H1-Stämmen offensichtlich. Es ist nicht ganz klar, inwiefern diese Ergebnisse durch die Einführung präziserer Diagnosewerkzeuge verzerrt wurden, die unser Labor entwickelt hatte: Der Nachweis von porzinem IAV und die Charakterisierung der Subtypen und Virusstämme von HA (H1av, H1hu, H1pdm, H3) und NA (N1av, N1pdm, N2) erfolgt durch drei RT-qPCRs in Echtzeit. Die erhöhte Sensitivität dieser Assays ermöglicht die direkte Analyse von Feldproben (insbesondere von Nasenabstrichen) unabhängig von der Virusisolierung in Zellkulturen (Henritzi et al. 2016). Diese Studien werden bis mindestens Ende 2017 weitergeführt.
Die kontinuierliche Überwachung der Schweinebestände in Europa ist unerlässlich, um die Entwicklung des porzinen IAV zu verfolgen. Wie sich beim Auftreten des H1N1-Virus 2009 zeigte, kann das porzine IAV eine wichtige Rolle bei der Entstehung neuer humaner pandemischer Viren spielen. Einblicke in die dynamischen Veränderungen der Genotypen und das Auftreten neuer Phänotypen mit möglicherweise erhöhten zoonotischen Neigungen von porzinem IAV ist das zentrale Thema der Politik von OneHealth.
Danksagungen
Aktuelle Studien zum porzinen IAV im Labor des Autors wurden von der IDT, Dessau (Deutschland), finanziert. Die Autoren danken Aline Maksimov und Carl Sell für die ausgezeichnete technische Unterstützung.



