Escherichia coli kommt natürlicherweise in der Darmflora von Schweinen vor und füht unter bestimmten Bedingungen zum Krankheitsbild der porzinen Colibacillose, einer Krankheit, die sich in unterschiedlichen Beschwerden manifestieren kann, zu denen u. a. neonatale Diarrhö, Diarrhö bei Absetzferkeln, die Ödemkrankheit, Sepsis etc. zählen.
Eine sorgfältige Diagnose beinhaltet eine ausführliche Anamnese, eine korrekte Auswahl der Proben und eine vollständige, von verschiedenen Verfahren unterstützte Differentialdiagnose. Der bloße Nachweis pathogener Stämme ist nicht immer ein Beweis für die Erkrankung, da häufig asymptomatische Träger gefunden werden. Die Identifizierung relevanter virulenter Stämme und deren Unterscheidung von anderer natürlicher Darmflora stellt eine hochaktuelle diagnostische Herausforderung dar. Und die Real-Time PCR (qPCR) zur Erkennung von Virulenzfaktoren (VF) von E. Coli erschien als ein wirkungsvolles Mittel, das nützliche Informationen liefern konnte.

Auf Grundlage der Extraktion von Nukleinsäuren aus verschiedenen biologischen Matrices (Kultur, Kot, Darm- oder rektaler Abstrich) erkennen qPCR-Assays die spezifischen genomischen Abschnitte von E. coli , die verschiedene VF codieren. In dieser Studie war jede Probe darauf ausgelegt, einen anderen VF (s. Tabelle 1) aufzufinden. Eine zusätzliche qPCR (ECCO) diente dazu, das Vorkommen von E. coli in den Proben zu bestätigen und sicherzustellen, dass alle Abschnitte dieses Verfahrens korrekt durchgeführt wurden. Die qPCR ist nicht nur ein qualitatives, sondern auch ein quantitatives Testverfahren (Abb. 1), was einen bedeutenden Vorteil gegenüber ihren Vorgängern, der konventionellen PCR darstellt.
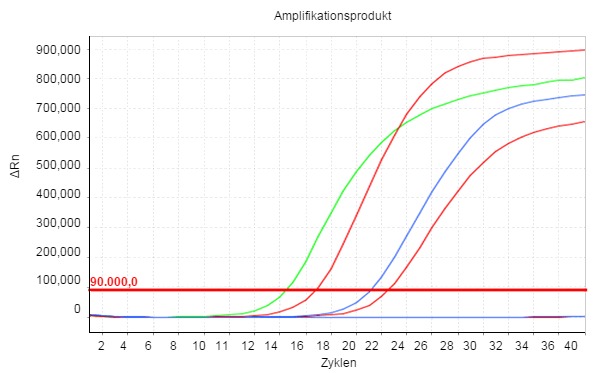
Abbildung 1. Das Amplifikationsprodukt wird in Echtzeit gezeigt, da mit der Entwicklung der Wärmezyklen das gemessene Fluoreszenzniveau (ΔRn) exponentiell ansteigt. Cq ist die Anzahl der Zyklen, bei denen man beginnt Fluoreszenz festzustellen. Je kleiner der Cq-Wert, desto höher ist die Anfangskonzentration des in der Probe untersuchten Parameters.
Die Bewertung der virulenten Stämme bei der Colibacillose erfolgte herkömmlicherweise durch eine anfängliche mikrobiologische Kultur, die folgende Isolierung von E. coli-Kolonien und die anschließende Beschreibung ihrer VF. Der wesentliche Nachteil dieser Methode ist, dass die Analyse nur bei einer beschränkten Anzahl ausgewählter Isolate durchgeführt werden kann, wobei die Genauigkeit der Diagnose auf der Annahme beruht, dass diese in der gesamten E. coli Population der untersuchten Probe repräsentativ sind. Dadurch besteht die Gefahr, Populationen von E. coli zu übersehen, die keine Mehrheiten darstellen, allerdings klinisch relevant sind.
Die qPCR bietet die Möglichkeit, die VF direkt an der klinischen Probe zu analysieren. Bei dieser Methode werden Zwischenkulturen und die jeweiligen Bestimmungen der verschiedenen ausgewählten Isolate vermieden, was zu beträchtlichen Zeit- und Kostensparnissen führt. Angesichts der quantitativen Natur des Verfahrens kann eine Beziehung zwischen der Anzahl an Kopien, die für ein spezifisches Virulenzgen gefunden wurden, und der gesamten E. coli-Population in der Probe festgestellt werden. Dadurch könnten wir die Möglichkeit der Isolierung eines Stamms mit einer besonderen Kombination von VF interpretieren.
Diese Methode unterliegt aufgrund der unbestimmten Zahl der Kopien der einzelnen Gene, die jeden VF innerhalb der verschiedenen Bakterienpopulationen codieren, gewissen Einschränkungen. Sie erfüllt aber die Zielsetzung, die Möglichkeit zu beurteilen, E. coli mit einer besonderen Kombination von VF in der Probe zu finden.
Ein praktisches Beispiel wird unten aufgeführt. In einem Fall eines klinischen Verdachts auf eine Ödemerkrankung wurde jeweils der Dünndarm von vier betroffenen Mastschweinen an das Labor geschickt um das Vorkommen Verotoxin bildender E. coli zu prüfen. Der Pool, der aus den 4 Dünndärmen bestand, wurde direkt durch qPCR ausgewertet und zeigte sehr ähnliche Cq-Werte für F18, Stx2e und ECCO (Tabelle 1). Diese Ergebnisse können als Population von E. coli interpretiert werden, die mit großer Mehrheit vorkommt und die Gene besitzt, die F18 und Stx2e, also einen Verotoxin bildenden Stamm, codieren. Ein leicht positiver Wert für F4 (Cq = 36,9) wurde auch erreicht, was auf ein sehr geringes Vorkommen von Populationen mit diesem VF hindeuten würde.
Gleichzeitig wurde eine mikrobiologische Kultur mit den Proben durchgeführt und 12 Kolonien von E. coli ausgewählt. Ein phänotypischer Vergleich zeigte das Vorkommen eines einzelnen Stamms (Abb. 2). Seine Charakterisierung durch die Bestimmung seiner VF durch qPCR bestätigte, dass es sich um einen potentiell Verotoxin bildenden Stamm handelte, da Gene nachgewiesen wurden, die F18 und Stx2e codieren.
Tabelle 1. qPCR-Ergebnisse.
| VF | Pool aus 4 Dünndärmen | Isolierter E. coli -Stamm |
| F4 | POS (Cq = 36.9) | neg |
| F5 | neg | neg |
| F6 | neg | neg |
| F18 | POS (Cq = 22.28) | POS (Cq = 16.58) |
| F41 | neg | neg |
| LT | neg | neg |
| STa | neg | neg |
| STb | neg | neg |
| Stx2e | POS (Cq = 22.39) | POS (Cq = 17.29) |
| eae | neg | neg |
| ECCO | POS (Cq = 21.1) | POS (Cq = 16.97) |
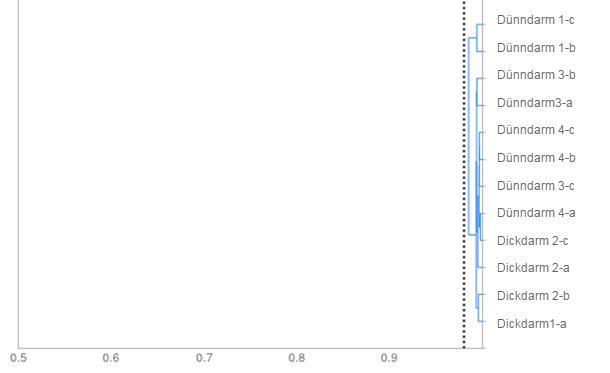

Abbildung 2. Dendrogramm: Aufgezeigt wird der Grad der Ähnlichkeit unter den Isolaten von E. coli entsprechend ihrer Form der Zuckerfermentation. Alle Isolate weisen eine Ähnlichkeit auf, die höher ist als der Schwellenwert, der bei 0,98 (punktierte Linie) gesetzt wurde, woraus wir schließen können, dass es sich um einen einzigen Stamm handelt.
Abschließend lässt sich sagen, dass die qPCR, die direkt bei klinischen Proben angewendet wird, eine erhebliche Einsparung von Zeit und Kosten darstellt und interpretationsfähige Informationen liefert, die Lösungen für die sensitive Diagnose der porzinen Colibacillose anbietet.


