Enterotoxische Escherichia coli (ETEC) sind die wichtigsten Krankheitserreger, die für die neonatale Diarrhö (ND), die Diarrhö bei Absetzferkeln (PWD) und die Ödemkrankheit (ED) bei Schweinen verantwortlich sind. Obwohl normalerweise junge Tiere während der Laktations- und Aufzuchtphase von der Colibacillose betroffen sind, können ETEC auch bei Mast- und Endmastschweinen gefunden werden. ETEC haften sich mit Fimbrien wie z. B. F4, F5, F6, F18 und F41 an die Mikrovilli des Dünndarms an und produzieren Enterotoxine, die lokal auf die Enterozyten wirken. Die ETEC-Enterotoxine werden aufgrund ihrer Hitzestabilität in die hitzelabilen Toxine LTa (eltA), LTb (eltB) und die hitzestabilen Toxine STa (estI), STb (estII) und EAST1 (astA) eingeteilt. Es wurde nachgewiesen, dass das Vorkommen von ETEC im Darm nicht ausreicht, um klinische Erkrankungen zu verursachen (Dewey CE et al, 1995). Die Pathogenese von ETEC ist komplex und zahlreiche Gene müssen reguliert werden, um Virulenzfaktoren zu produzieren, zu aktivieren und abzusondern. Umweltsignale wie z. B. die Temperatur, die Ionenkonzentration, Fe++, der pH-Wert und die O2-Konzentration können die Expression dieser Gene regulieren (Nagy B et al, 2005) und könnten verschiedene Gene für Virulenzfaktoren an- oder abschalten. Darüber hinaus könnten sich zahlreiche Faktoren der Tierhaltung, einschließlich Futterumstellung, frühes Absetzen und ständige Fluktuation auf das Auftreten klinischer Formen von ETEC auswirken.
In dieser Studie untersuchten wir die Prävalenz von ETEC-positiven Schweinen in Betrieben ohne klinische Symptome von Diarrhoe und das Muster der Antibiotikaresistenz, das bei Isolaten von ETEC-Stämmen beobachtet wurde. Wir führten eine Querschnittsstudie durch, an der 11 Betriebe teilnahmen, bei denen keine klinischen Symptome oder eine frühere Erkrankung an Colibacillose vorlagen. Die untersuchten Betriebe wurden aufgrund eines der folgenden Kriterien ausgewählt: Größe der Sauenbestände (225 ± 30) und zur Schlachtung gelieferte Endmastschweine/Jahr (mindestens 2.500 verkaufte Endmastschweine/Jahr). Die ausgewählten Betriebe hatten dieselbe Bewirtschaftungsform und waren alle Aufzucht- und Mastbetriebe. Von jedem Betrieb wurden 90 rektale Abstriche genommen, um das Vorkommen von eltA, estI oder beiden und die folgende Prävalenz verschiedener Genotypen in jeder Produktionsphase festzustellen. Von jeder Produktionsphase wurden 30 zufällig ausgewählte Schweine wie folgt ausgewählt: Laktation (Saugferkel, 21 ± 3 Tage alt); Aufzucht (Mastferkel, 86 ± 3 Tage alt); Endmast (Endmastschweine, 165 ± 3 Tage alt). Schweine, die als ETEC-positiv (ETEC+) eingestuft wurden, waren diejenigen, bei denen eltA, estI oder beide Gene nachgewiesen wurden.

Prävalenz der Gene eltA und estI bei Schweinen ohne Diarrhö
Aus insgesamt 990 untersuchten Proben wurden 150 Proben (15,2% – 95,0% CI ± 2,24) positiv auf das Vorhandensein von eltA, estI oder beiden getestet. Von diesen 150 ETEC+-Schweinen waren jeweils 82,7% (95,0% CI ± 6,05), 11,3% (95,0% CI ± 5,07) und 6,0% (95,0% CI ± 3,8) positiv auf estI, eltA bzw. estI/eltA . Der Gesamtanteil an ETEC+-Tieren erhöhte sich von 16,6% (95,0% CI ± 5,95) in der Laktationsphase auf 66,0% (95,0% CI ± 7,58) in der Aufzuchtphase. Der prozentuale Anteil der Schweine, die Überträger waren, ging bei den Endmastschweinen schlagartig auf 17,3% (95,0% CI ± 6,05) zurück. In 10 der 11 beurteilten Betriebe gab es ETEC+-Schweine. Unter den 150 ETEC+-Schweinen wurden 40 ETEC-Stämme (26,7%) (95,0% CI ± 13,71) aus 30 Proben von Mastferkeln (75,0%) (95,0% CI ± 13,42), 8 Proben von Endmastschweinen (20,0%) (95,0% CI ± 12,4) und 2 Proben von Saugferkeln (5,0%) (95,0% CI ± 6,75) isoliert.
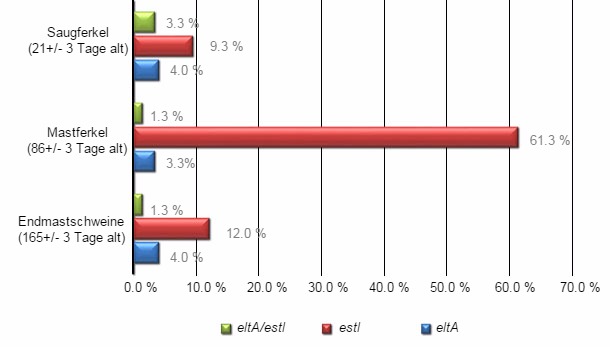
Abbildung 1: Prävalenz von eltA (Toxin LT) und estI (Toxin STa) Genen, die im Bereich des konfluenten Wachstums auf Trypton-Soja-Bouillon (TSB) festgestellt wurde.
Von jedem Betrieb wurden insgesamt 90 rektale Abstriche genommen, um das Vorkommen von eltA (Toxin LT) und estI (Toxin STa) Genen und die folgende Prävalenz verschiedener Genotypen in jeder Produktionsphase zu bestimmen. Untersucht wurden 30 aufgrund ihres Alters und der Produktionsphase zufällig ausgewählte Schweine der 3 verschiedenen Phasen (Laktation: Saugferkel, 21 ± 3 Tage alt; Aufzucht: Mastferkel, 86 ± 3 Tage alt; Endmast: Endmastschweine, 165 ± 3 Tage alt).
Antibiotikaempfindlichkeit von ETEC-Stämmen
Zwei der 40 ETEC-Stämme reagierten empfindlich auf alle untersuchten Antimikrobiotika. Alle Stämme reagierten empfindlich auf Amikacin (AMK), Colistin (CST), Cefotaxim (CTX) und Cefoxitin (CXT). 38 Stämme (95%) waren gegenüber mindestens einem untersuchten Antimikrobiotikum resistent und all diese Stämme zeigten eine Resistenz gegenüber Tetracyclin (TET). Festgestellt wurden 18 verschiedene Profile antimikrobieller Resistenz und bei 52,5 % der Stämme eine Multiresistenz (MAR) gegenüber mehr als drei Antibiotika (Abb. 2).
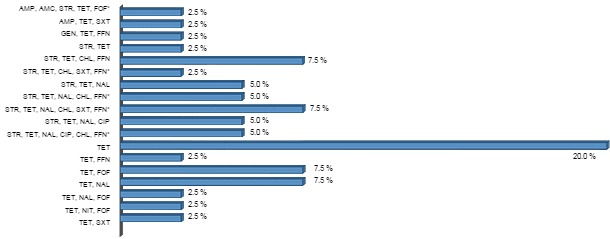
Abbildung 2: Das Profil antimikrobieller Resistenz der ETEC-Stämme bei Schweinen ohne Diarrhö
Bei 40 ETEC+-Isolaten wurden 18 verschiedene Muster multipler Antibiotikaresistenz beobachtet. Der Anteil jedes Multiresistenzmusters wird mit den horizontalen Balken dargestellt. Ampicillin (AMP), Cephalothin (CET), Amoxicillin/Clavulansäure (AMC), Gentamicin (GEN), Streptomycin (STR), Tetracyclin (TET), Nalidixinsäure (NAL), Ciprofloxacin (CIP), Chloramphenicol (CHL), Florfenicol (FFN), Cotrimoxazol (SXT), Nitrofurantoin (NIT), Fosfomycin (FOF). * = Multiple Antimikrobiotikaresistenzen

Bei dieser Studie stellten wir fest, dass zahlreiche ETEC-Genotypen in Schweinebeständen zusammen vorkommmen und zirkulieren, ohne dass in den verschiedenen Produktionsstufen eine klinische Manifestation der neonatalen Diarrhö, der Diarrhö bei Absetzferkeln oder der Ödemkrankheit auftritt. Darüber hinaus wurden zahlreiche antibiotikaresistente Profile bei ETEC-Isolaten von nicht klinisch erkrankten Tieren beobachtet. Die gewonnenen Informationen sind nicht nur für diagnostische und epidemiologische Zwecke von Bedeutung, sondern auch für das Verständnis der Dynamik und Ökologie von ETEC bei Schweinen in verschiedenen Produktionsphasen, die eine mögliche Ursache von klinischer Colibacillose darstellen können.


