Die – nicht ganz so neuen – Herausforderungen, wenn es um das PRRS-Virus geht
Es ist keine Neuigkeit, dass das Virus des Porzinen Reproduktiven und Respiratorischen Syndroms (PRRSV) wichtige Eigenschaften aufweist, die zum ständigen Wiederauftreten der Krankheit in Schweinebeständen beitragen, was zu erheblichen Problemen bei der Tiergesundheit und wirtschaftlichen Verlusten führt. Eine dieser Eigenschaften ist die Fähigkeit, schnell zu mutieren und potenziell schwerwiegender zu werden und/oder sogar Tiere zu infizieren, die zuvor „immun“ gegen eine andere PRRSV-Variante waren. Um einen in einem Betrieb auftretenden PRRSV-„Typ“ zu verstehen – egal ob es sich um ein Virus aus einem neuen Ausbruch oder ein „residentes“ Virus handelt – ist es daher allgemein üblich geworden, das PRRSV-Genom zu analysieren.
Dies geschieht in der Regel durch die Sequenzierung eines wichtigen Teils von PRRSV, des Gens ORF5, um zu herauszufinden, wo das Virus evolutionär einzuordnen ist, und es mit früheren Viren zu vergleichen, die in diesem Betrieb oder in der Nachbarschaft/Region nachgewiesen wurden.

PRRSV-Sequenzinformationen wurden in der Praxis weitgehend genutzt, um:
- zu verstehen, welches Virus die Probleme verursacht
- Hinweise zur Ausbruchsquelle zu erhalten
- zwischen Impfstoffstämmen und „residenten“/neu eingeschleppten Stämmen zu unterscheiden
- Impfstoffe auszuwählen usw.
In jüngster Zeit wird zur Virusklassifikation zunehmend die Klassifikation nach „Linien“ verwendet. Diese Methode wurde um das Jahr 2010 entwickelt (Shi et al., 2010), als PRRSV in 9 verschiedene Linien unterteilt wurde, und weiter verfeinert, als PRRSV weiter in Sublinien unterteilt wurde.
Der „Elefant im Raum“: Reicht die Sequenzierung einer einzigen Probe aus?
Die PRRSV-Sequenzierung ist in der Regel nicht billig, daher wird sie eher in „besonderen Situationen“ und nicht routinemäßig durchgeführt (z. B. bei neuen Ausbrüchen, nach Virusinokulationen usw.). Außerdem wird in diesen Fällen in der überwiegenden Mehrheit der diagnostischen Fälle nur eine Probe oder ein Proben-Pool (von vielen, die üblicherweise im landwirtschaftlichen Betrieb entnommen werden) für die Sequenzierung verwendet.
Dies bedeutet, dass Tierärzte, Erzeuger und andere Mitarbeiter im Bereich der Tiergesundheit in vielen Fällen nur eine einzige Sequenzinformation verwenden, um wichtige Rückschlüsse auf die Quelle des Virus zu ziehen (insbesondere bei neuen Ausbrüchen), die Ausbruchsuntersuchungen zu steuern und über künftige Maßnahmen zu entscheiden.
Wir vermuteten, dass wir angesichts der schnellen Mutationsrate von PRRSV und der Größe moderner Schweinebetriebe innerhalb einer Probenahme mehrere PRRSV-Linien finden könnten, was bedenklich wäre. Eine grafische Darstellung des Grundprinzips unserer Studie ist in Abb. 1 zu sehen.
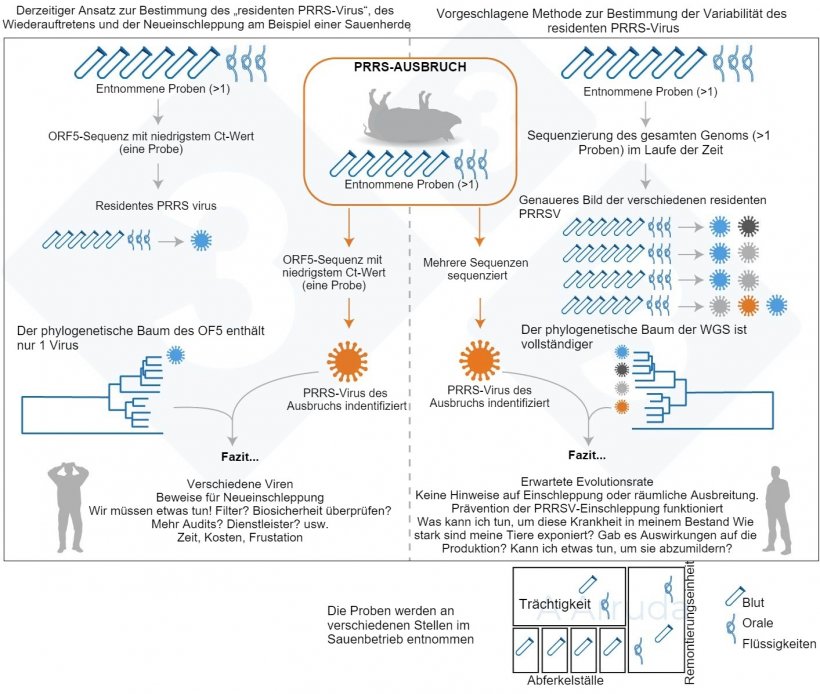
Abb. 1: Illustration eines Beispiels für den derzeit üblichen Ansatz zur Bestimmung der PRRSV-Variante in einem Betrieb (linkes Feld) und die von uns vorgeschlagene Methode zur Bestimmung der Variabilität des residenten PRRS-Virus (rechtes Feld)
Untersuchung der Frage, wie viele PRRSV-Stämme wir in unseren Betrieben finden können
Wir nahmen fünf Betriebe in die Studie auf, 3 Zuchtbetriebe und 2 Endmastbetriebe, und entnahmen etwa ein Jahr lang monatlich verschiedene Proben (Tonsillenabstriche, orale Flüssigkeiten, Gewebesäfte). Die Probenahmestellen in den Ställen waren immer dieselben und waren räumlich über das gesamte Gelände verteilt, um eine möglichst hohe Repräsentativität für den gesamten Betrieb zu gewährleisten. Jeden Monat entnahmen wir bis zu 16 Proben pro Standort, die alle mittels quantitativer PCR getestet wurden; anschließend wurden die positiven Proben ORF5-sequenziert.
Was haben wir herausgefunden?
Unsere wichtigste Erkenntnis war, dass wir unter Feldbedingungen bei einer einzigen Probenahme in Zuchtbeständen bis zu drei und in Mastschweinbeständen bis zu zwei verschiedene PRRSV-Linien nachweisen konnten. Dies ist in Abb. 2 dargestellt.
Abb. 2: PRRSV-Sublinien, die in den Untersuchungsbetrieben (1-5) während der Dauer unserer Probenahmen (in Monaten) in den verschiedenen Probenarten nachgewiesen wurden.
| Probenahme | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Betrieb | Probenart | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 1 | Gewebesaft | L1H | L1H | ||||||||||
| Tonsillenabstrich | |||||||||||||
| 2 | Gewebesaft | L1H | L1H | L1A | L1H | L1H | |||||||
| L1H | |||||||||||||
| L8† | |||||||||||||
| Tonsillenabstrich | L1H | ||||||||||||
| 3 | Gewebesaft | L1H | L1H | L1H | L1H | ||||||||
| Tonsillenabstrich | |||||||||||||
| 4 | Gewebesaft | L5† | L1A | ||||||||||
| L5† | |||||||||||||
| Tonsillenabstrich | L1A | L5† | L5† | L1A | |||||||||
| L5† | |||||||||||||
| 5 | Gewebesaft | L1A | L1A | L5† | |||||||||
| Tonsillenabstrich | L1A | L1A | |||||||||||
Wir stellten auch fest, dass in unserer Studie die Sequenzierung der oralen Flüssigkeiten sehr schwierig und die der Gewebesaftproben am einfachsten war. Da es sich in beiden Fällen um Sammelproben handelte, lässt sich dies möglicherweise durch insgesamt höhere Ct-Werte erklären, die auf eine geringere Virusmenge in oralen Flüssigkeiten als in den Gewebesaftproben hinweisen. Wir versuchten es mit der WGS, konnten aber letztendlich von keiner unserer Proben vollständige Genomsequenzen erhalten. Wir vermuten, dass dies auf die Art der von uns verwendeten Proben zurückzuführen sein könnte, müssen aber noch weitere Nachforschungen zu diesem Thema anstellen.
Diese Ergebnisse zeigen, wie wichtig es ist, bei bedeutsamen Entscheidungen die Sequenzierung mehrerer Proben in Betracht zu ziehen, auch wenn uns bewusst ist, dass dies mit höheren Kosten verbunden ist.
Danksagungen
Das National Pork Board hat dieses Projekt finanziell unterstützt. Ebenso möchten wir uns bei den Tierärzten und Erzeugern bedanken, die uns bei der Probenahme unterstützt haben.






