Die Häufigkeit bakterieller und viraler Erreger bei Saugferkeldiarrhoe - Ergebnisse eines europäischen Monitoringprogramms
11-Jul-2022 (vor 2 Jahre 3 Monate 22 Tage)
Durchfallerkrankungen sind eine der Hauptursachen für neonatale Mortalität, verringerte Gewichtszunahmen und einen hohen Medikamenteneinsatz in Schweinebeständen. Die Haupterreger sind Bakterien wie Escherichia coli (E. coli) und Clostridium perfringens (C. perfringens), sowie Rota- und Coronaviren.
Eine Studie basierend auf dem Probenmaterial eines europäischen Monitoringprogramms untersuchte das Vorkommen dieser Erreger im Kot von an Neonataler Diarrhoe (ND) erkrankten Saugferkeln.
Material und Methoden
Alle in der Studie verwendeten Proben stammten von Saugferkeln. Insgesamt wurden 324 (vorwiegend Kot-)Proben aus 116 Betrieben kulturell auf E. coli, C. perfringens und Clostridioides difficile (C. difficile) untersucht. Proben aus 79 dieser Betriebe wurden zusätzlich mittels PCR auf Rotavirus A und C sowie auf das Porzine Epizootische Diarrhoe (PED) und das Transmissible Gastroenteritis Virus (TGEV) getestet. Dadurch bestand die Studie aus zwei Analysen, basierend auf den Unterschieden im untersuchten Erregerspektrum in den Betrieben. In der ersten Analyse (1) wurde die Häufigkeit der bakteriellen Erreger in allen Proben der 116 Betriebe ermittelt. Die zweite Analyse (2) untersuchte das gemeinsame Auftreten von mehr als einem Erreger mittels der kombinierten Datensätze zu Bakterien und Viren in den 79 Betrieben.
Ein Großteil der 116 Betriebe (Abb. 1) lag in Deutschland (DE; n=59), gefolgt von den Niederlanden (NL; n=19) und Polen (PL; n=19), Dänemark (DK; n=9), Großbritannien (UK; n=8), Österreich (AU; n=1) und Irland (IR; n=1).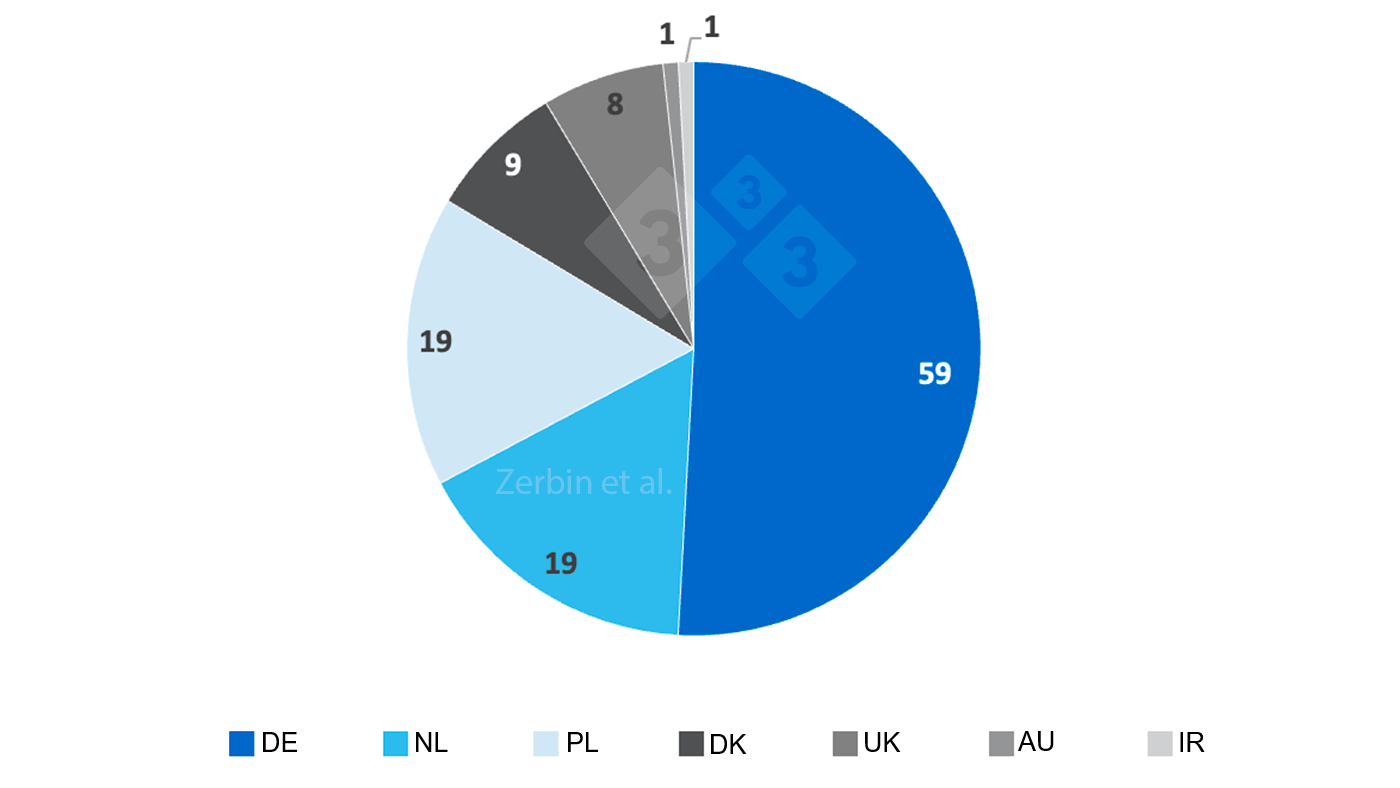
Die E. coli und C. perfringens Isolate wurden in beiden Analysen zum Nachweis virulenzassoziierter Gene molekularbiologisch typisiert, um ihr Virulenzpotential zu beurteilen (Strutzberg-Minder and Dohmann 2015). Zusätzlich wurde der semi-quantitative bakterielle Keimgehalt für E. coli, Cl. perfringens und Cl. difficile in Analyse 1 ermittelt, abhängig von ihrem kulturellen Wachstum (Tab. 1).
Ergebnisse
Analyse 1:
In der bakteriologischen Untersuchung aller Proben wurden insgesamt 710 Isolate nachgewiesen (Abb. 2). Dabei wurden E. coli am häufigsten isoliert (48,6%), gefolgt von C. perfringens (33,9%) und C. difficile (15,9%). Das semi-quantitativen Keimwachstum (Tab. 1) lag bei 99,4% der E. coli und 96,7% der Cl. perfringens Isolate im mittel- bis hochgradigen Bereich. Dagegen wurde bei Cl. difficile nur mittelgradiges (46,9%) bis geringgradiges Wachstum (53,1%) nachgewiesen. Nur 16% der E. coli Isolate zeigten Hämolyse (Tab. 1), und auch unter denen mit definiertem Pathotyp bzw. potentiell virulenten Stämmen waren nur 16% der E. coli hämolysierend. Dies bestätigt, dass die Eigenschaft „Hämolyse“ als alleiniges Merkmal zu Identifizierung von pathogenen E. coli nicht sinnvoll ist.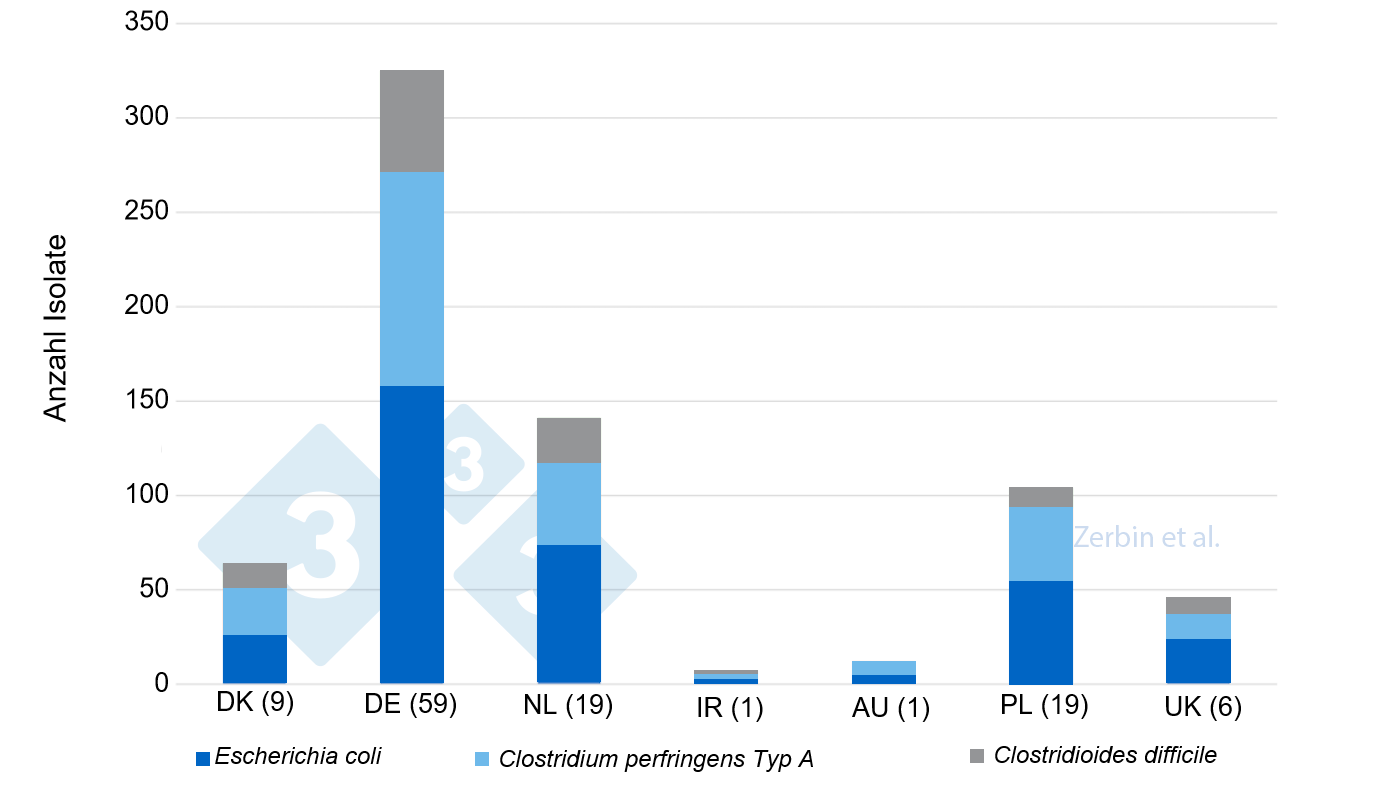
Tabelle 1: Anzahl (n) der kulturell nachgewiesenen Bakterienisolate in Analyse 1, sowie Anteile des semi-quantitativen Nachweises innerhalb jeder Bakterienspezies.
| Erreger | n | % | ||
|---|---|---|---|---|
| hochgradiger Gehalt | mittelgradiger Gehalt | Geringgradiger Gehalt | ||
| Escherichia coli | 345 | 77.1 | 22.3 | 0.6 |
| davon hämolysierend | 57 (16.5%) | |||
| Clostridium perfringens | 241 | 46.9 | 49.8 | 3.3 |
| Clostridioides difficile | 113 | 0.0 | 46.9 | 53.1 |
Nach der Typisierung und Zuordnung zu den jeweiligen Betrieben blieben 276 E. coli und C. perfringens Isolate übrig. Diese unterschieden sich hinsichtlich ihrer virulenzassoziierten Gene bzw. Toxingene innerhalb einer Farm. Basierend auf diesen Virulenzgenen konnten 19,9% der E. coli Isolate einem der mit ND assoziierten Pathotypen zugeordnet werden (EDEC, EPEC, ETEC, NTEC). Weitere 104 Isolate (37,7%) trugen verschiedene Gene für Fimbrien und Adhäsine sowie Toxine und wurden daher als potentiell virulent eingestuft. Somit wurden insgesamt 57,6% (n=159) der E. coli Isolate als (potentiell) virulent eingestuft. Die Abb. 3 stellt die Verteilung der Typisierungsergebnisse für E. coli bezogen auf die Ursprungsländer dar.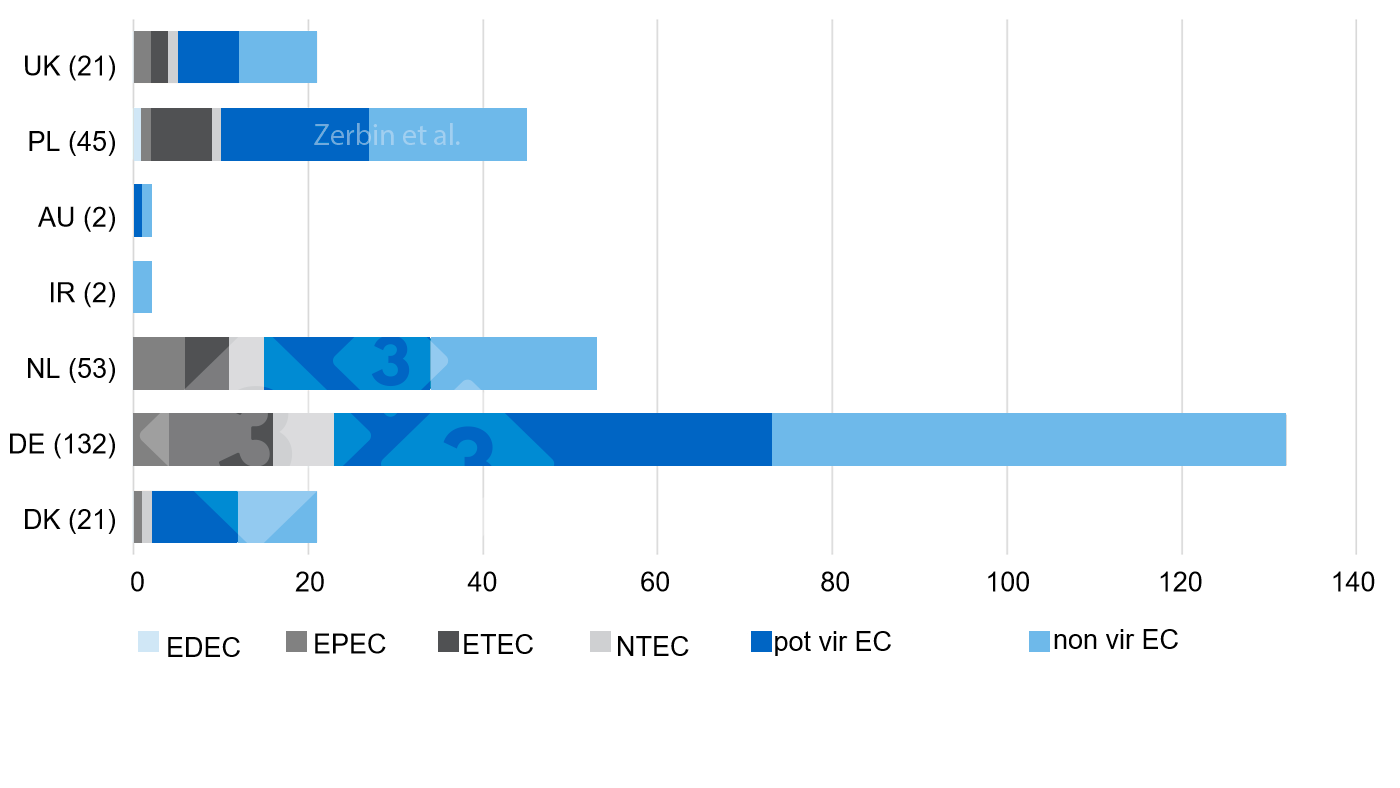
Alle C. perfringens Isolate konnten durch die Typisierung C. perfringens Typ A (CPA) zugeordnet werden. Von ihnen trugen 90,6% (n=106) sowohl die kodierenden Gene für das α- als auch das ß2-Toxin und zeigten somit ein hohes Virulenzpotential. Die übrigen Isolate (9,4%, n=11) wiesen lediglich das α-Toxingen auf (Abb. 4).
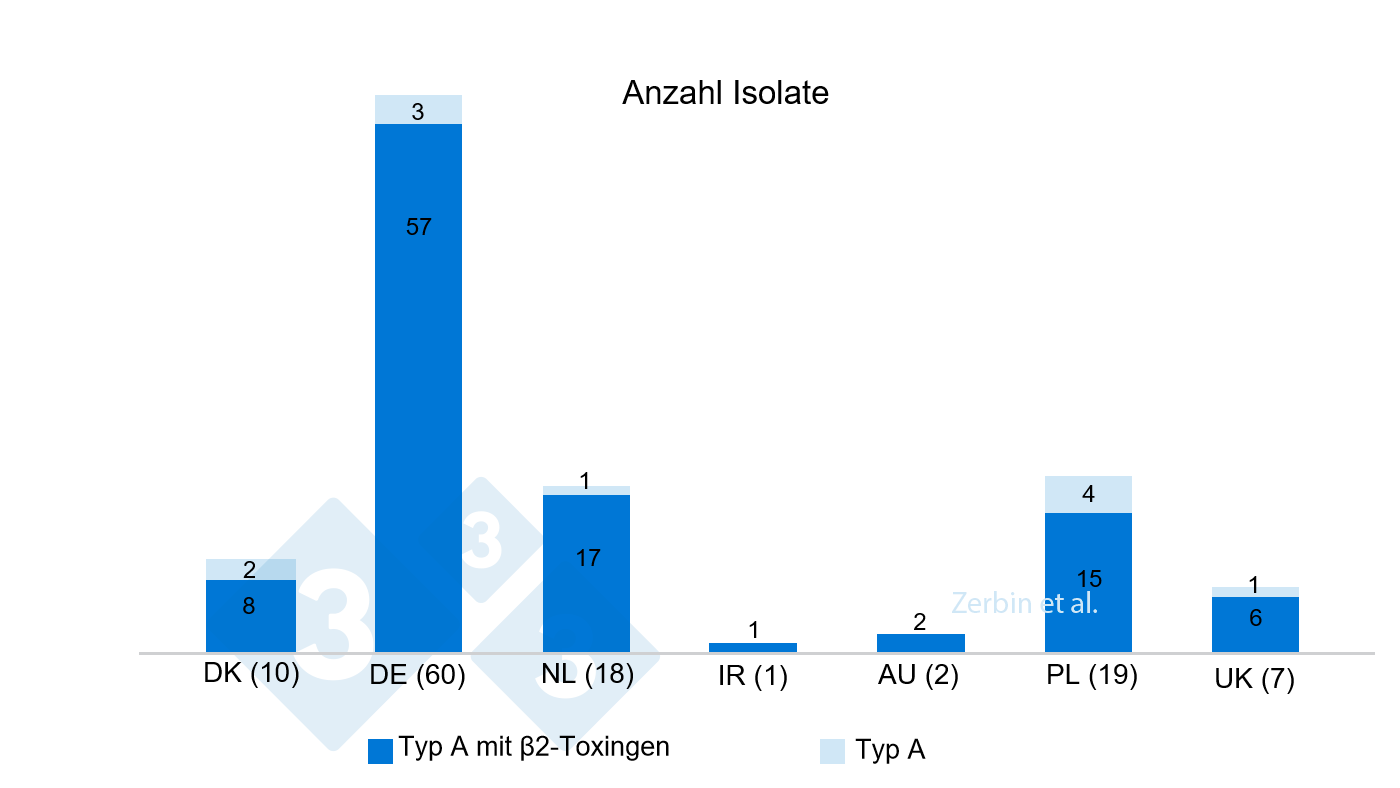
Analyse 2:
In der Untergruppe mit 79 Betrieben waren die am häufigsten nachgewiesenen Erreger E. coli (96,2%), C. perfringens (87,3%), Rotavirus (74,7%) und C. difficile (46,8%). PEDV wurde in zwei Betrieben nachgewiesen, TGEV in keinem der Betriebe. Daher wurde die Gruppe der Coronaviren in der weiteren Analyse nicht berücksichtigt.
Die anschließende Typisierung ergab einen Anteil virulenter oder potentiell virulenter E. coli Isolate von 78,9% (n=60), und C. perfringens Typ A mit ß2-Toxingen von 81,0% (n=64) in den Betrieben.
In 25,3% (n=20) der Betriebe wurden alle vier verschiedenen Erreger zugleich nachgewiesen, in 39,2% (n=31) wurden drei Erreger gefunden, zwei Erreger in 31,6% (n=25), und in 3,8% (n=3) der Betriebe wurde nur einer der Erreger detektiert. Nur in einem Betrieb konnte keiner der genannten Erreger für ND nachgewiesen werden.
Die Kombination aus E. coli mit Rotavirus und CPA wurde dabei am häufigsten gefunden (n=19/ 24,1%). Die Kombination aus E. coli, CPA und Cl. difficile kam deutlich seltener vor (n=8).
E. coli als der am häufigsten nachgewiesener ND Erreger wurde zumeist mit CPA mit ß2-Toxingen (64,4%) oder Rotaviren (64,6%) nachgewiesen.
Diskussion
Die vorliegende Studie untersuchte das Vorkommen relevanter Erreger der ND in europäischen schweinehaltenden Betrieben, welche freiwillig an einem Monitoringprogramm für Saugferkeldurchfall teilgenommen hatten. Aufgrund der Verzerrung der Probenauswahl durch das Studiendesign sollte die Interpretation der Studienergebnisse unter leichtem Vorbehalt erfolgen. Die Betriebe nahmen freiwillig aufgrund von Problemen mit Saugferkeldurchfall teil. Nichtsdestotrotz zeigen die vorliegenden Daten klare und realistische Trends hinsichtlich der ND verursachenden Erreger, die bereits in der Literatur beschrieben wurden.
Beide Analysen der Studie bestätigen E. coli und C. perfringens als Haupterreger bei Saugferkel-Durchfall. Die Ergebnisse zeigten hier, dass knapp 58% der E. coli Isolate und knapp 91% der C. perfringens Isolate als virulent oder potentiell virulent anzusehen sind. In der zweiten Analyse wurden relevante E. coli und C. perfringens Isolate in ca. 80% der Betriebe gefunden.
Ein interessantes Ergebnis ist der sehr geringe Nachweis von Coronaviren (PEDV und TGEV) in dem Probenmaterial der Studie. Auch wenn sie als mögliche Verursacher der ND Erwähnung finden, ist ihr Nachweis in der Literatur meistens gering, und durch TGEV verursachter Durchfall ist in Europa selten geworden.
Die kombinierte Analyse der bakteriellen und viralen Erreger in den 79 Betrieben ergab den gleichzeitigen Nachweis von mindestens zwei Erregern in 95% der Betriebe. Dies verdeutlicht, dass ND beim Schwein selten eine monokausale Ursache hat. Das sollte im Hinblick auf prophylaktische und therapeutische Ansätze berücksichtigt werden.
